Víctor Lorente Leal1; Pilar Pozo Piñol2; Javier Bezos Garrido1,3; Julio Álvarez Sánchez1,3; Bernat Pérez de Val4, Christian Gortázar Schmidt2; Lucía de Juan Ferré1,3; Beatriz Romero Martínez1,3
1. Centro de Vigilancia Sanitaria Veterinaria, Universidad Complutense de Madrid.
2. Grupo SaBio, Instituto de Investigación en Recursos Cinegéticos, Consejo Superior de Investigaciones Científicas & Universidad de Castilla-La Mancha.
3. Departamento de Sanidad Animal, Facultad de Veterinaria, Universidad Complutense de Madrid.
4. Centre de Recerca en Sanitat Animal, Institut de Recerca i Tecnología Agroalimentàries (IRTA-CReSA).
Tuberculosis humana – un viejo conocido
El día 24 de marzo es una fecha señalada; en 1882, tal día como hoy, el Dr. Robert Koch anunciaba el descubrimiento del “bacilo tuberculoso” o Mycobacterium tuberculosis, la bacteria causante de la tuberculosis (TB). Para muchas personas esta enfermedad infecciosa suena a algo lejano y propio de las películas o novelas victorianas. Sin embargo, la realidad es bien distinta: la TB es posiblemente una de las enfermedades infecciosas más relevantes para el ser humano. Con excepción de la irrupción temporal de la COVID-19 durante la pandemia, la TB es, con diferencia, la principal causa de mortalidad por un agente infeccioso a nivel mundial de nuestro tiempo. Sólo en 2021, alrededor de 10,5 millones de personas contrajeron la infección y aproximadamente 1,6 millones de personas murieron como consecuencia de ésta.
A diferencia de la COVID-19, que lleva con nosotros unos pocos años, la TB es una enfermedad muy antigua que ha atormentado al ser humano desde los albores de la historia. Se han encontrado indicios de TB en restos humanos del neolítico con hasta 9.000 años de antigüedad, y hay descripciones de la enfermedad en escrituras de varias civilizaciones antiguas, como la mesopotámica, la egipcia o la griega. Pero no fue hasta hace 141 años que se puso el foco en el verdadero culpable, lo que supuso el punto de inflexión necesario para que comenzásemos a plantearnos eliminar esta enfermedad de nuestras vidas.
Tras este trasfondo histórico nos situamos en 2014, año en el que 194 países de la Asamblea Mundial de la Salud adquirieron el compromiso de acabar definitivamente con la pandemia mundial de TB en 2030. Este programa, denominado “End TB” o “Poner Fin a la TB”, puso sobre la mesa varios objetivos para reducir el impacto de la TB a nivel mundial, como por ejemplo reducir el número de muertes por TB en un 90% con respecto al número de muertes acontecidas en el año 2015 (es decir, en 1,6 millones de personas).
Pero eliminar esta enfermedad será una tarea más ardua y costosa de lo inicialmente anticipado. Entre 2015 y 2019, la reducción en el número de muertes por TB estuvo por debajo de las expectativas, al situarse en torno al 17% (de 1,8 millones a 1,5 millones de personas). Y en 2021 la disrupción producida por la pandemia por la COVID-19 fue tal que los servicios relacionados con la prevención, el diagnóstico y tratamiento de la TB se vieron drásticamente mermados. Lejos de desmoralizarnos, la tendencia observada hasta la llegada de la COVID-19 debería recordarnos que las medidas implantadas durante estos años sí que han tenido un efecto directo sobre la vida de las personas, y debería animarnos a seguir “remando todos a una” para superar este bache y conseguir alcanzar la meta establecida hace ocho años.

Sello distribuido por la administración postal de la República Democrática de Alemania en 1982 donde se muestra la efigie de Robert Koch y el artículo científico sobre la “Etiología de la TB”, que publicó tres semanas después de su descubrimiento.
Tuberculosis zoonósica – más allá de la frontera humana
Una de las muchas razones que hacen que poner fin a la TB sea una tarea titánica es que, aunque no lo parezca, esta enfermedad no es un problema exclusivo de la especie humana. De las once “especies” de bacilos tuberculosos que comprenden el complejo Mycobacterium tuberculosis (CMTB) tan sólo dos están perfectamente adaptadas al ser humano (M. tuberculosis y M. africanum). El resto están adaptadas en mayor o menor medida a distintas especies o grupos de animales; por ejemplo, M. bovis es el agente principal de la TB en el ganado bovino, pero también puede infectar a muchas otras especies animales, mientras que M. pinnipedii se ha aislado principalmente de mamíferos marinos, especialmente pinnípedos como focas o leones marinos, pero también de delfines.
Las miembros del CMTB que afectan a los animales, especialmente M. bovis, también pueden afectar al ser humano, y viceversa, por lo que la TB es una enfermedad zoonósica de gran importancia para la salud humana. Los últimos datos publicados por la Organización Mundial de la Salud, datados de 2017, estimaron que en 2016 hubo 147.000 nuevos casos de TB zoonósica y aproximadamente 12.500 muertes debido a esta enfermedad. Al igual que su vertiente humana, la TB zoonósica tiene mayor peso en aquellas regiones del mundo más desfavorecidas, especialmente en el continente africano y en el sudeste asiático. Además, debido a la ausencia de programas y métodos diagnósticos capaces de identificar de manera rutinaria la especie del CMTB que causa la TB en personas, es bastante probable que la proporción de infectados por M. bovis esté subestimada. Por esta razón, controlar la infección en las poblaciones animales es crucial para eliminar la TB de las poblaciones humanas.
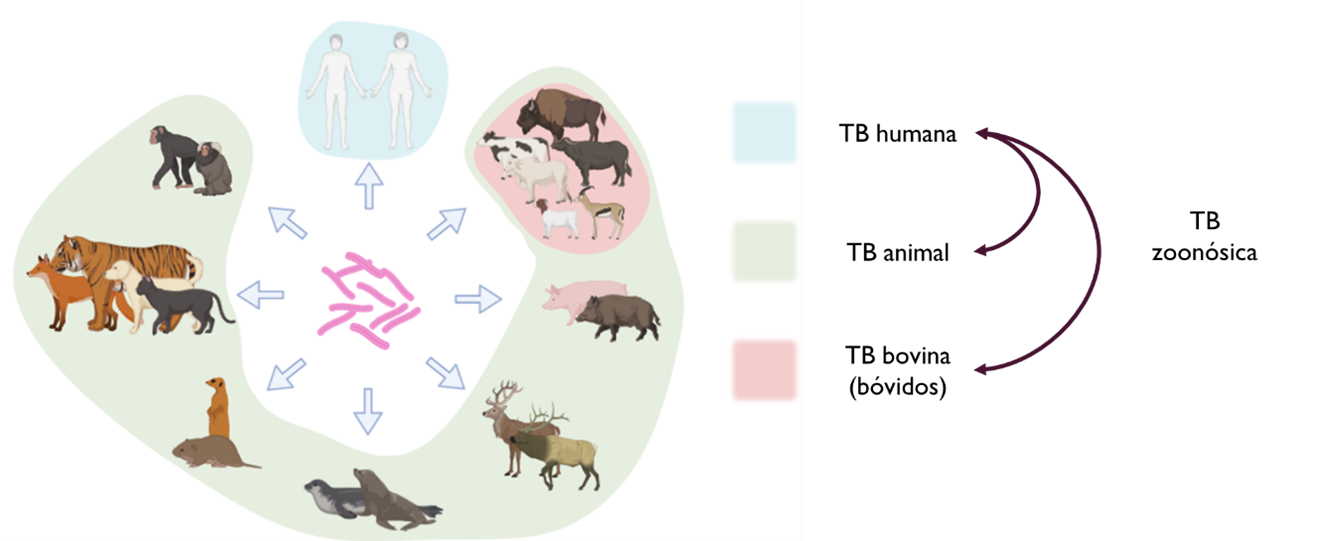
La TB es una enfermedad infecciosa muy compleja que afecta a múltiples especies de mamíferos, pudiendo transmitirse entre humanos (TB humana), animales (TB animal) y entre ambos (TB zoonósica).
Tuberculosis animal – una realidad muy compleja
Hablar de TB en animales no es una tarea sencilla, ya que las propias definiciones que se han empleado para describir la enfermedad han variado con el paso de los años. Aunque en tiempos de su descubrimiento la TB era una enfermedad relevante en mamíferos, el uso del término “TB animal” no se ha generalizado hasta hace pocos años. Tradicionalmente, la TB en animales era considerada una enfermedad asociada al ganado bovino y producida por M. bovis, por lo que el concepto de TB bovina se usaba de manera generalizada para referirse a esta infección en mamíferos no humanos. Debido a la gran importancia que tiene la producción y aprovechamiento de los bóvidos en las economías de todo el mundo, y debido a que el consumo de leche de vaca sin pasteurizar era una de las principales vías de transmisión de la TB zoonósica, la mayoría de las estrategias de control y erradicación de la infección en distintas regiones del mundo se enfocaron hacia al ganado bovino y la higiene alimentaria. De esta manera, el éxito en la eliminación de la infección a través de los programas de erradicación de la TB bovina, basados en el diagnóstico y sacrificio de los animales infectados, así como la reducción de los casos de TB zoonósica en aquellos países donde se implantaban medidas de higiene en los productos lácteos, ayudó a generalizar la concepción de que la TB bovina y la TB animal eran conceptos intercambiables, aunque su repercusión y control no lo fueran.
Pero el paso del tiempo y la mejora en las técnicas empleadas para detectar y caracterizar los bacilos tuberculosos presentes en distintas especies animales nos demostró que la realidad no era tan sencilla como pensábamos. Por un lado, se identificaron múltiples variantes del CMTB que podían infectar al ganado bovino, como M. caprae o M. orygis, y, por otro, se reveló la capacidad de M. bovis de mantenerse en gran variedad de nichos ecológicos, dando lugar a ciclos de infección que involucran a múltiples especies domésticas y silvestres. Así, por ejemplo, la TB animal en la Península Ibérica puede encontrarse principalmente en el ganado bovino, pero en determinadas regiones la infección puede darse también en la fauna silvestre, especialmente en jabalíes y ciervos. Por esta razón, parece más conveniente pensar en “comunidades de mantenimiento” en lugar de hospedadores concretos como reservorios de infección.
Por esta razón, abordar el problema únicamente desde la perspectiva del ganado bovino sería inútil, razón por la cual actualmente se aboga por una definición más global de la enfermedad y por la implementación de medidas de control en las poblaciones domésticas, silvestres y en la interfaz entre estas. Si se añade a esta ecuación la relevancia de la TB zoonósica anteriormente mencionada, puede verse porqué la lucha contra la TB es un desafío que ejemplifica el concepto “One Health” o “Una sola salud”.
Epidemiología molecular de la TB animal - ¿y tú de quién eres?
Para poder controlar y eliminar una enfermedad de una población primero es necesario conocer su distribución, frecuencia y magnitud, así como los distintos factores que determinan su aparición y persistencia. Estos atributos forman el cimiento de la epidemiología, una disciplina que estudia la salud de las poblaciones y sus cambios. Ya hemos visto en anteriores párrafos que la realidad de la TB es algo compleja, por lo que el estudio de su epidemiología no nos va a resultar nada sencillo, especialmente si tenemos en cuenta un rasgo bastante peculiar de este grupo de patógenos: que, a pesar de infectar a distintos grupos de mamíferos, estos microorganismos son prácticamente idénticos.
El estudio de brotes se basa en la identificación de los cambios genéticos (o mutaciones) presentes en los aislados obtenidos en las personas o animales infectados, estableciéndose el grado de relación entre éstos en función del número de mutaciones presentes en sus genomas (el material genético completo de un organismo). Es decir, cuantas menos mutaciones tengan dos aislados, más probable es que estén relacionados entre sí (por ejemplo, que procedan de la misma granja de origen). Pero, al contrario de lo que sucede con otros patógenos, los miembros del CMTB no sólo comparten el 99,9% de su genoma, sino que además tienen una capacidad muy limitada de experimentar mutaciones genéticas. Aunque esto resulta muy interesante, especialmente si tenemos en cuenta que estas bacterias llevan con nosotros miles de años, resulta un problema importante a la hora de estudiar el origen de los brotes que producen. Debido a su limitada capacidad de mutación, establecer el origen de una infección puede ser una tarea muy complicada, ya que es posible que los aislados de una misma especie de bacilo tuberculoso obtenidos en años distintos sean idénticos entre sí.

La epidemiología molecular nos permite detectar aquellas mutaciones genéticas que diferencian a los aislados de bacilos tuberculosos, de tal manera que somos capaces de establecer si existe una relación entre ellos (bacilos negros frente a bacilo azul) que pudiera indicar una fuente común de infección (contacto entre fauna salvaje y ganado bovino).
Desde los años 90 las diferencias entre aislados de miembros del CMTB eran detectadas principalmente mediante dos técnicas moleculares que analizan una porción muy pequeña de su genoma: el espoligotipado y el análisis del número variable de repeticiones en tándem (VNTRs por sus siglas en inglés). El uso de estas técnicas supuso una revolución, ya que nos permitió diferenciar los distintos miembros del CMTB y conocer, en cierta manera, cómo de variadas eran las distintas poblaciones de bacilos tuberculosos. En el caso de M. bovis, el uso del espoligotipado nos permitió descubrir que este patógeno se dividía en grupos genéticos, conocidos como complejos clonales, que estaban más o menos distribuidos por el mundo. En el caso de España, el complejo clonal más frecuente es el Eu-2, que también lo es en Portugal y puede encontrarse en otros países como Brasil. Por otro lado, los VNTRs nos permitieron conocer la variabilidad existente dentro de los grupos genéticos descubiertos mediante el espoligotipado, lo cual tuvo una gran repercusión a la hora de hacer estudios epidemiológicos en TB animal. Estas técnicas no sólo nos permitieron conocer si existía un parentesco entre aislados de la misma o distintas explotaciones ganaderas, en el caso de la TB bovina, sino que también nos permitieron establecer posibles relaciones entre aislados procedentes de distintas especies animales (por ejemplo, entre el ganado bovino y la fauna silvestre en los alrededores de la explotación).
A pesar de su éxito, llegó un punto en el que la utilidad de estas técnicas se vio limitada. En determinados contextos, estas técnicas no eran lo suficientemente discriminatorias como para diferenciar entre aislados de distintos brotes. Por suerte para nosotros, los avances tecnológicos nos han permitido desarrollar técnicas mucho más resolutivas, como es el caso de la secuenciación masiva de genomas.
La secuenciación masiva y la TB – hasta el nucleótido y más allá
Los genomas de todos los seres vivos están compuestos por unas moléculas, conocidas como nucleótidos, que solemos codificar con cuatro letras (las famosas A, C, G y T de la película GATTACA). La secuenciación masiva de genomas es un proceso que engloba una serie de técnicas y herramientas informáticas mediante las cuales somos capaces de conocer la composición y el orden de secuencia de estas letras en el genoma de un organismo. Comparando la secuencia de nucleótidos de distintos aislados de bacilos tuberculosos somos capaces de identificar mutaciones puntuales entre estas por el cambio de un solo nucleótido (SNP por sus siglas en inglés), o si hay nucleótidos que han sido insertados (inserciones) o eliminados (deleciones) del genoma. Teniendo en cuenta que las mutaciones puntuales se acumulan con el paso del tiempo, su número y localización en el genoma nos puede ayudar a reconstruir la evolución de estos patógenos.
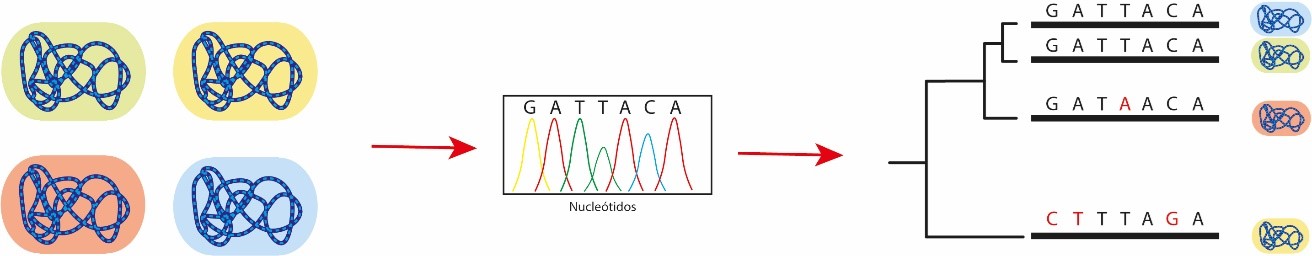
La secuenciación masiva nos permite reconstruir la secuencia del genoma bacteriano hasta el nivel del nucleótido. Comparando la secuencia de distintos aislados de una misma especie podemos establecer su parentesco de una manera mucho más fiable que mediante otras técnicas. En este diagrama, los bacilos verde y azul son más parecidos entre sí en comparación con el resto.
Hasta hace un par de décadas tan sólo éramos capaces de conocer la secuencia de letras de unas pequeñas porciones del genoma, y conseguirlo en genomas completos era algo extremadamente costoso, complejo y limitado a unos pocos organismos. Con la mejora y el abaratamiento de los procesos, hoy en día es relativamente asequible secuenciar un genoma, especialmente aquellos tan pequeños como los de los bacilos tuberculosos, lo cual ha popularizado cada vez más su uso en el ámbito de la TB, tanto humana como animal. Las aplicaciones de esta tecnología son infinitas y, a día de hoy, son varios los miembros de la Red RISA que realizan estudios de secuenciación masiva en TB animal, no sólo con M. bovis sino también con otras especies como M. caprae.
La secuenciación masiva de genomas se puede emplear para determinar de manera rápida cual es la diversidad existente en las poblaciones de bacilos tuberculosos a unos niveles de detalle hasta ahora inalcanzables. En función de la diversidad existente en un área o región concreta, o lo similar que es ésta con respecto a otras regiones de su entorno, se pueden establecer todo tipo de hipótesis que expliquen la evolución de este patógeno. En 2021, el IRTA-CReSA publicó un estudio en el que se analizaron 125 aislados de M. bovis, procedentes de ganado bovino y caprino, así como jabalíes, de Cataluña entre los años 2008-2018. La secuenciación masiva de genomas reveló una gran diversidad genética de los aislados de M. bovis en esta CCAA, con hasta siete grupos genéticos distintos, que habría pasado desapercibida si únicamente se hubiesen empleado las técnicas tradicionales. Esto podría reflejar que este patógeno lleva circulando en nuestro país durante muchos siglos, con múltiples introducciones en el tiempo. Curiosamente, al comparar los aislados de Cataluña con otros países, los investigadores vieron que algunos se relacionaban con los obtenidos en Reino Unido, Francia, EEUU, México e incluso Sudáfrica, lo cual podría asociarse con relaciones comerciales en el pasado.
En 2022, el Centro VISAVET realizó el primer estudio de secuenciación masiva a gran escala, centrándose en este caso en el tercer grupo genético más frecuente en nuestro país de acuerdo a la técnica de espoligotipado. En él se analizó la diversidad genética de 136 aislados de M. bovis procedentes de ganado bovino y fauna salvaje (principalmente jabalí y ciervo) de una amplia variedad de regiones de España entre 2005 y 2018. A pesar de que todos los aislados formaban en principio parte del mismo grupo genético (espoligotipo), se detectó una gran diversidad genética. Los investigadores demostraron que los aislados tuberculosos eran normalmente más similares entre sí cuando procedían de la misma región geográfica, independientemente de la especie animal de la que hubiesen sido obtenidos. Esto podría confirmar el largo recorrido de la TB en España, donde M. bovis ha estado circulando entre las poblaciones de bovino y especies silvestres durante extensos periodos de tiempo, y refleja el complejo sistema ecológico en el que se mantiene esta enfermedad en nuestro país. Curiosamente, los aislados procedentes de fauna silvestre fueron menos diversos que aquéllos recuperados de bovino, lo cual podría reflejar que, en el caso del ganado, las infecciones podrían haber partido de múltiples fuentes (por ejemplo, a través de movimientos entre explotaciones, pastos comunales, etc.).
Al permitirnos establecer relaciones de parentesco mucho más cercanas, la secuenciación masiva también nos permite determinar rápidamente cuál es el origen más probable de un brote o cómo se transmiten los bacilos tuberculosos entre diferentes especies animales. En 2020, investigadores del IRTA-CReSA realizaron una investigación epidemiológica a partir de un caso de TB (en este caso, por M. caprae) en un jabalí del Parque Natural de Montseny, en la provincia de Barcelona. Para investigar la extensión del brote, se analizaron muestras de jabalí durante cinco temporadas de caza consecutivas entre los años 2015 y 2020, así como muestras de cabras y ganado bovino de explotaciones que se encontrasen en o lindasen con el parque natural. Se identificaron tres aislados de M. caprae, procedentes de jabalíes cazados en 2015 y 2016, casi idénticos a los de una cabra de una explotación de caprino de carne localizada en el mismo parque, y que sufrió varios casos de TB en 2017. En este caso, la hipótesis más plausible fue que la infección podría haberse dado por el contacto indirecto entre los jabalíes y las cabras en los pastos comunales que existen dentro del parque natural. Curiosamente, los aislados de jabalí que se encontraron en el parque fueron asimismo muy distintos a los obtenidos en otra explotación de cabras de leche situada en las inmediaciones de Montseny que también tuvo TB en 2017, lo cual descartaba que estos últimos pudiesen formar parte del mismo brote.
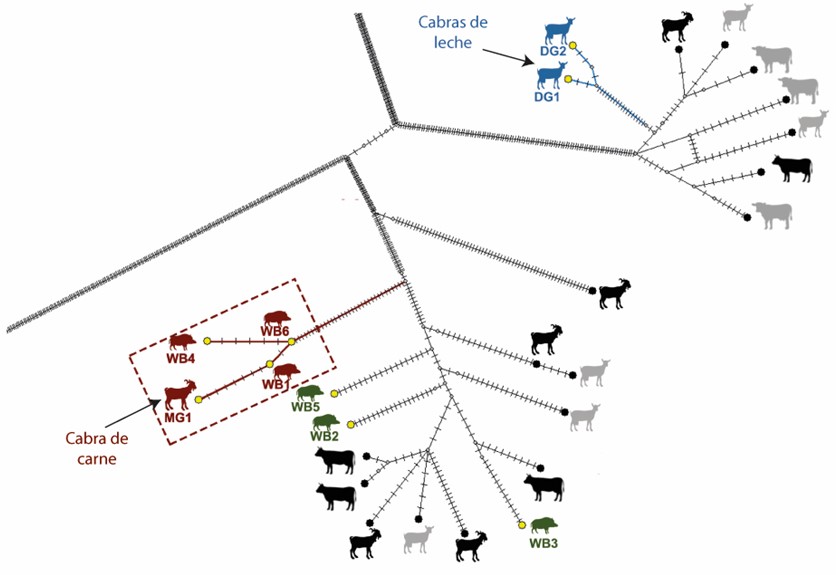
La secuenciación masiva de genomas de mostró la cercanía genética entre varios aislados de M. caprae obtenidos en una cabra de carne y tres jabalíes (rojo) del Parque Natural de Montseny y demostró que estos casos no estaban relacionados con un brote de TB caprina en una explotación de leche en los alrededores del parque natural (azul). Imagen adaptada del artículo de investigación original.
El nivel de detalle que nos ofrece la secuenciación tiene su aplicación más allá de la TB animal, y puede ser de gran utilidad en casos de TB zoonósica: no sólo nos permitiría conocer la relación entre aislados de animales y humanos, sino que nos permitiría buscar si existen mutaciones que puedan hacer más peligrosas para el ser humano a las bacterias causantes de la enfermedad, ya sea porque tengan una mayor virulencia o porque sean resistentes a los antibióticos que usamos para tratarlas. En el año 2021, investigadores de múltiples centros de investigación de España, incluyendo al IRTA-CReSA, la Universidad Autónoma de Barcelona y el Centro VISAVET, describieron un caso de TB zoonósica poliresistente en un ganadero en Cataluña. Normalmente M. bovis es resistente de manera natural al antibiótico pirazinamida, que es uno de los antibióticos de primera línea que se emplean para tratar la TB en humanos. En este caso, el aislado de M. bovis era además resistente a la isoniazida, otro antibiótico de primera línea en TB humana, pero también a la etionamida, un antibiótico de segunda línea habitualmente empleado para tratar cepas multirresistentes de M. tuberculosis. Al tratarse de un ganadero que trabajaba con ganado bovino y pequeños rumiantes, la investigación epidemiológica llevó a analizar a los animales con los que había estado en contacto. Los resultados del estudio demostraron que el aislado del ganadero era casi idéntico a los de dos ovejas con las que había tenido contacto, lo cual reforzó la hipótesis de una adquisición zoonósica de la infección. Por otro lado, los investigadores pudieron identificar aquellas mutaciones que conferían la resistencia a los antibióticos, demostrando una vez más el potencial de la secuenciación masiva, que podría usarse para hacer un seguimiento de este tipo de características en los bacilos tuberculosos que circulan en los animales en España.
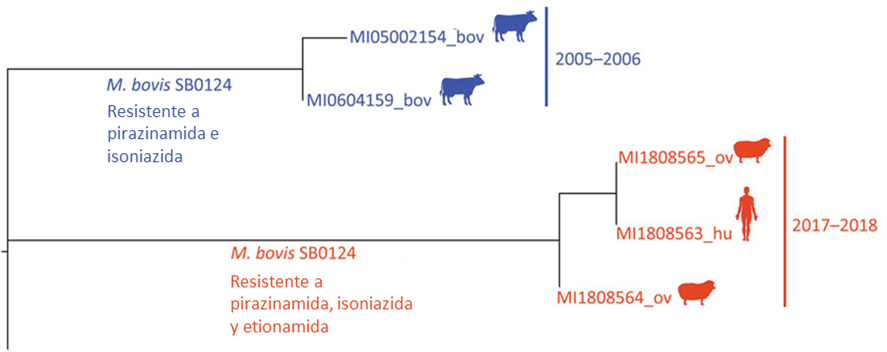
La secuenciación masiva demostró que el aislado de M. bovis poliresistente procedente de un ganadero era casi idéntico a los aislados obtenidos de sendas ovejas (naranja) con las que había tenido contacto previo, y no con otros aislados resistentes obtenidos de vacas más de diez años atrás (azul). Imagen adaptada del artículo de investigación original.
La secuenciación masiva al servicio de la TB
El uso de la secuenciación masiva para estudiar la TB está despegando en nuestro país y en muchas otras partes del mundo, por lo que todavía tenemos mucho trabajo por delante y será necesario establecer sinergias y desarrollar nuevas plataformas que integren toda la información genómica que se está generando. Llegados a este punto no es difícil ver la gran variedad de aplicaciones y el valor que nos ofrece esta tecnología. Poner la secuenciación masiva al servicio del estudio de la TB sin duda mejorará nuestra capacidad de estudiar esta enfermedad infecciosa, ayudándonos a definir mucho mejor las causas detrás de los brotes de TB. De esta manera, la secuenciación masiva puede ser un gran apoyo al Programa Nacional de Erradicación de la Tuberculosis Bovina, permitiendo introducir nuevas medidas con las que proteger la salud de los animales y, en definitiva, del ser humano. Esperemos que, con el tiempo, el uso de esta tecnología se limite a estudiar la TB como un resto arqueológico y no el problema sanitario que actualmente es.

Uso de imágenes
Las imágenes empleadas en este artículo de divulgación han sido obtenidas de las siguientes fuentes de acceso libre:
Wikimedia Commons
Photo Credit:Content Providers(s): CDC/Dr. George Kubica, Public domain, via Wikimedia Commons
